XVIII Congreso Nacional de Biotecnología y Bioingeniería
Identificación mediante el gen recA y espectrometría de masas de L. paraplantarum productora de compuestos antimicrobianos de orígen proteíco
Jessica J. Hurtado Ríos, Israel García-Cano, Julio César Almanza Pérez y Edith Ponce-Alquicira.
Gen recA
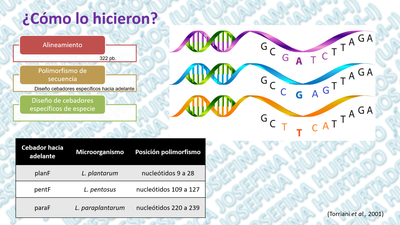
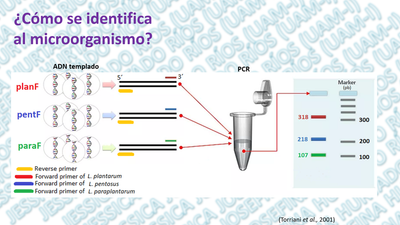
Sin embargo, cuando las especies que tienen un 70% o más de similitud de ADN generalmente tienen más del 97% de identidad de secuencias de RNAs; por lo tanto, la identidad de las secuencias de ARNr 16S puede no ser suficiente para garantizar que dos taxones pertenezcan a la misma especie, por lo que el análisis de la secuencia de ADN ribosómico 16S no es el método apropiado para reemplazar la reasociación del ADN para delinear especies y medir las relaciones intraespecíficas. Una alternativa para diferenciar especies con una similitud de ADN mayor al 97% es el uso de del gen recA y la proteína RecA como marcadores filogenético. Rec A es una proteína pequeña (352 aminoácidos en Escherichia coli) implicada en la recombinación de ADN homóloga, la inducción de SOS y la mutagénesis inducida por daño en el ADN. Estas funciones implican múltiples actividades bioquímicas, incluida la unión al ADN (doble y monocatenario), el emparejamiento e intercambio de ADN homólogo, la hidrólisis del ATP y la eliminación coproteolítica de las proteínas LexA, IcI y UmuD. Debido a su papel fundamental, el gen recA es omnipresente, y su producto genético ha sido propuesto como un marcador filogenético para especies emparentadas lejanamente (Torriani et al., 2001; Bringel et al., 2005).
Espectrometría de masas


La espectrometría de masas conocida como matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF MS), es una técnica que permite la identificación de microorganismos mediante el análisis de proteínas, principalmente ribosómicas, a través de la creación de un espectro de masas que es específico para cada especie. Un microorganismo dado presentará siempre una serie de picos característicos en el espectro, y esto permite la creación de bases de datos con los espectros de masas que presentan los distintos microorganismos. El espectro obtenido para un determinado microorganismo se compara automáticamente con la base de datos, y el resultado se emite junto a un puntaje o score. Ésta técnica, consta de cuatro pasos: la recuperación de una colonia aislada, la realización de un espectro de masas, la comparación con la base de datos y la entrega de resultados (Relloso et al., 2015).
La muestra obtenida de la colonia aislada del microorganismo en estudio que se aplica en la placa de metal pulido consta de metodologías diferentes: análisis de células intactas o transferencia directa, y extracción previa de proteínas. Para el análisis de células intactas, se selecciona la colonia de interés y se aplica directamente sobre la placa de metal pulido; mientras que para la extracción con etanol-ácido fórmico, se resuspende la colonia de interés con agua bidestilada y etanol, centrifugando y eliminando el etanol, para posteriormente añadir ácido fórmico al 70% y la misma cantidad de acetonitrilo para la disrupción de la pared celular. Una vez centrifugada la muestra, se recoge el sobrenadante y se aplica en la placa de metal pulido. A continuación, para cualquiera de las metodologías empleadas, se cubre la muestra con la solución matriz (generalmente una solución saturada de ácido alfaciano-4-hidroxicinamico, acetonitrilo y ácido trifluoroacético) la que se cristaliza al dejarse secar a temperatura ambiente. Esta matriz tiene dos funciones fundamentales; primero expone las proteínas intracelulares mediante la ruptura de la membrana celular y segundo, facilita la vaporización y la ionización de las proteínas mediante un haz de laser pulsante. Una vez ionizadas estas proteínas viajan por una cámara de vacío siendo detectadas al final. Dependiendo de la relación masa/carga de cada fragmento será el tiempo que ésta demore en llegar al final. Este "tiempo de vuelo" es utilizado para construir el espectro específico de las masas. La manera en que este espectro es analizado va a depender del software utilizado para realizar la identificación. Este espectro es comparado automáticamente con un archivo y el resultado es entregado automáticamente (García et. al., 2012, Jordana-Luch et al., 2012).
Una vez obtenido el espectro de masas y comparado con los existentes en la base de datos, el software le adjudica una identificación y un valor indicativo de la fiabilidad de dicha identificación. En el caso de Biotyper, se emplea una escala logarítmica que va de 0 a 3, y se recomienda dividir el rango en tres intervalos: un valor ≥2 indica una identificación fiable a nivel de especie, un valor entre 1.7 y 2 indica un parentesco muy cercano y ofrece una identificación fiable a nivel de género, y un valor <1,7 ofrece una identificación poco fiable (Jordana-Luch et al., 2012).
La muestra obtenida de la colonia aislada del microorganismo en estudio que se aplica en la placa de metal pulido consta de metodologías diferentes: análisis de células intactas o transferencia directa, y extracción previa de proteínas. Para el análisis de células intactas, se selecciona la colonia de interés y se aplica directamente sobre la placa de metal pulido; mientras que para la extracción con etanol-ácido fórmico, se resuspende la colonia de interés con agua bidestilada y etanol, centrifugando y eliminando el etanol, para posteriormente añadir ácido fórmico al 70% y la misma cantidad de acetonitrilo para la disrupción de la pared celular. Una vez centrifugada la muestra, se recoge el sobrenadante y se aplica en la placa de metal pulido. A continuación, para cualquiera de las metodologías empleadas, se cubre la muestra con la solución matriz (generalmente una solución saturada de ácido alfaciano-4-hidroxicinamico, acetonitrilo y ácido trifluoroacético) la que se cristaliza al dejarse secar a temperatura ambiente. Esta matriz tiene dos funciones fundamentales; primero expone las proteínas intracelulares mediante la ruptura de la membrana celular y segundo, facilita la vaporización y la ionización de las proteínas mediante un haz de laser pulsante. Una vez ionizadas estas proteínas viajan por una cámara de vacío siendo detectadas al final. Dependiendo de la relación masa/carga de cada fragmento será el tiempo que ésta demore en llegar al final. Este "tiempo de vuelo" es utilizado para construir el espectro específico de las masas. La manera en que este espectro es analizado va a depender del software utilizado para realizar la identificación. Este espectro es comparado automáticamente con un archivo y el resultado es entregado automáticamente (García et. al., 2012, Jordana-Luch et al., 2012).
Una vez obtenido el espectro de masas y comparado con los existentes en la base de datos, el software le adjudica una identificación y un valor indicativo de la fiabilidad de dicha identificación. En el caso de Biotyper, se emplea una escala logarítmica que va de 0 a 3, y se recomienda dividir el rango en tres intervalos: un valor ≥2 indica una identificación fiable a nivel de especie, un valor entre 1.7 y 2 indica un parentesco muy cercano y ofrece una identificación fiable a nivel de género, y un valor <1,7 ofrece una identificación poco fiable (Jordana-Luch et al., 2012).
Bibliografía
•Torriani, S., Felis, G.E. and Dellaglio, F. (2001). Differentiation of Lactobacillus plantarum, L. pentosus, L. paraplantarum by recA gene sequence analysis and multiplex PCR assay with recA gene-derived primers. Applied and Environmental Microbiology. 67. 3450–3454.
•Bringel, F., Castioni, A., Olukoya, D.K., Felis, G.E., Torriani, S., and Dellaglio F. (2005) Lactobacillus plantarum subsp. argentoratensis subsp. nov., isolated from vegetable matrices. International Journal of Systematic and Evolutionary Microbiology. (55): 1629– 1634.
•Bourassa L, Butler-Wu SM. (2015) MALDI-TOF mass spectrometry for microorganism identification. In: Tang AS, Y-W, ed. Methods in Microbiology, vol 42. Current and Emerging Technologies for the Diagnosis of Microbial Infections. Cambridge, MA: Academic Press: 37–85.
•Jordana-Lluch, E.; Martró Català, E. y Ausina Ruiz V. (2012) La espectrometría de masas en el laboratorio de microbiología clínica. Enfermedades Infecciosas y Microbiología Clínica 30. 635–44.
•Bringel, F., Castioni, A., Olukoya, D.K., Felis, G.E., Torriani, S., and Dellaglio F. (2005) Lactobacillus plantarum subsp. argentoratensis subsp. nov., isolated from vegetable matrices. International Journal of Systematic and Evolutionary Microbiology. (55): 1629– 1634.
•Bourassa L, Butler-Wu SM. (2015) MALDI-TOF mass spectrometry for microorganism identification. In: Tang AS, Y-W, ed. Methods in Microbiology, vol 42. Current and Emerging Technologies for the Diagnosis of Microbial Infections. Cambridge, MA: Academic Press: 37–85.
•Jordana-Lluch, E.; Martró Català, E. y Ausina Ruiz V. (2012) La espectrometría de masas en el laboratorio de microbiología clínica. Enfermedades Infecciosas y Microbiología Clínica 30. 635–44.
Contacto
- Manhattan, Nueva York, NY, Estados Unidos
- 985-245-1730
- Ejemplo@ejemplo.com
- Lunes a viernes - 8:00-19:00
